【TBDig】CS224W 10.1-Deep Generative Models for Graphs
作者:互联网
CS224W 10.1-Deep Generative Models for Graphs
视频地址,课件和笔记可见官网。
【油管英字】CS224w 斯坦福图网络机器学习2019_哔哩哔哩 (゜-゜)つロ 干杯~-bilibili
http://web.stanford.edu/class/cs224w/

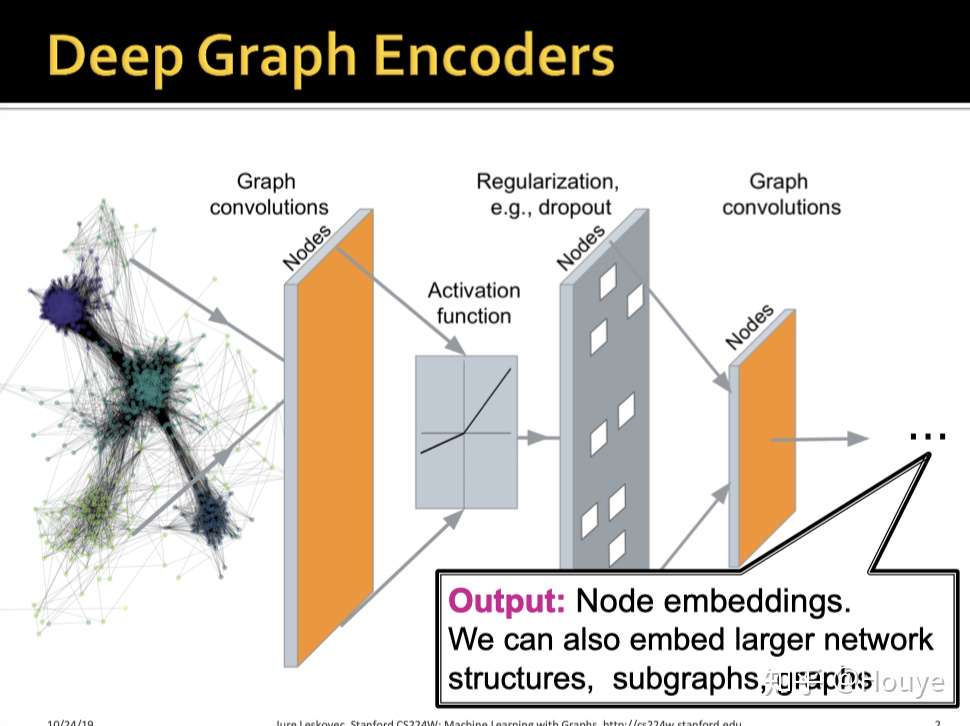
这里的graph encoder指的是将图上的信息进行编码的NN,包括图卷积等模型不是特指GAE.
虽然GNN可以应用于各种任务,节点级别/图级别, CV, NLP等等. 但是,GNN的核心没有变,就是如何学习节点表示,即node embedding.
GNN和普通NN最大的区别就充分挖掘了图上的结构信息(节点的邻接情况),而不是只是像传统NN一样对属性信息进行处理.
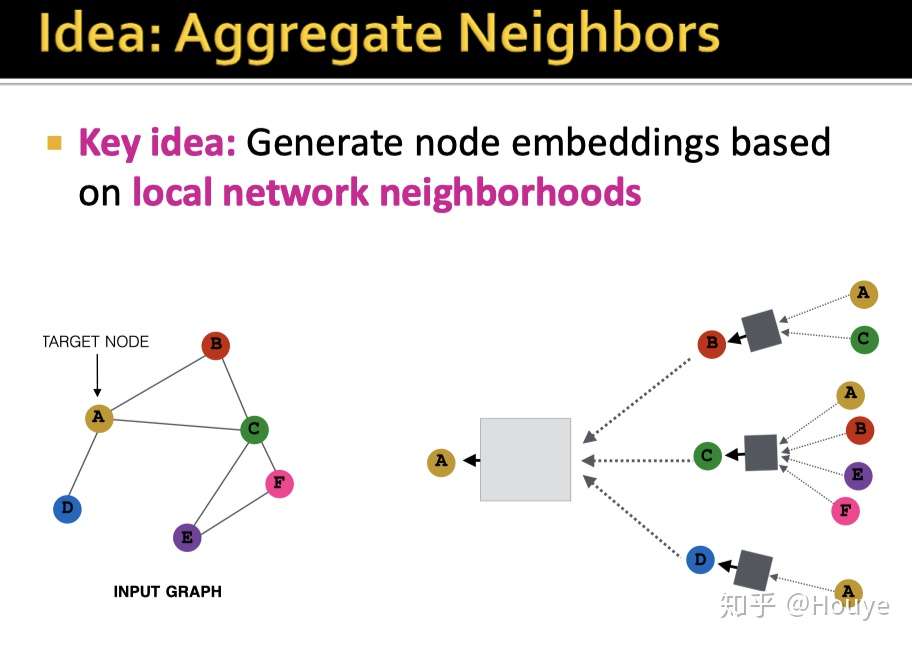
这里作者将节点A的邻居进行了整理展开.BCD都是直接与A一跳相连的节点,就是一阶邻居.
同样的,B的一阶邻居有AC.
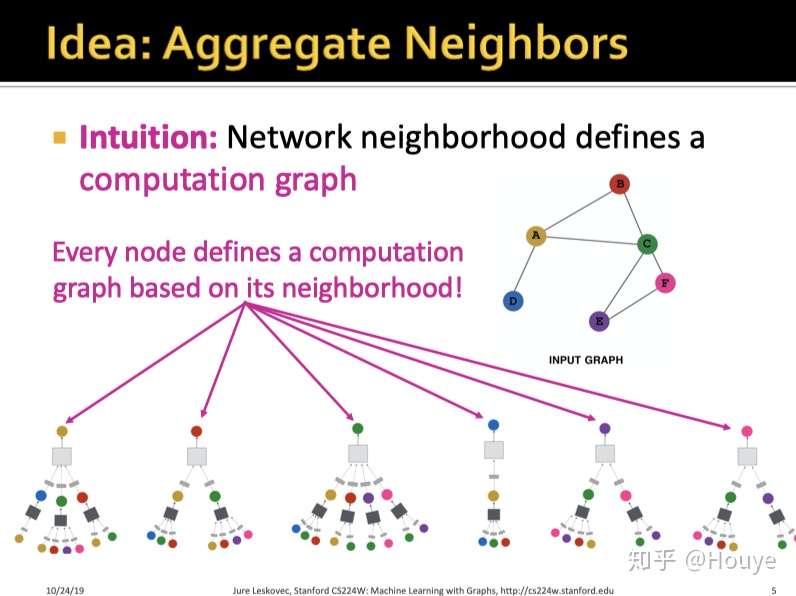
类比A的邻居展开,我们可以得到图上所有节点的邻居展开.
这里要展开成这种样子呢? 是为了方面我们后续实现GNN的邻居聚合过程.
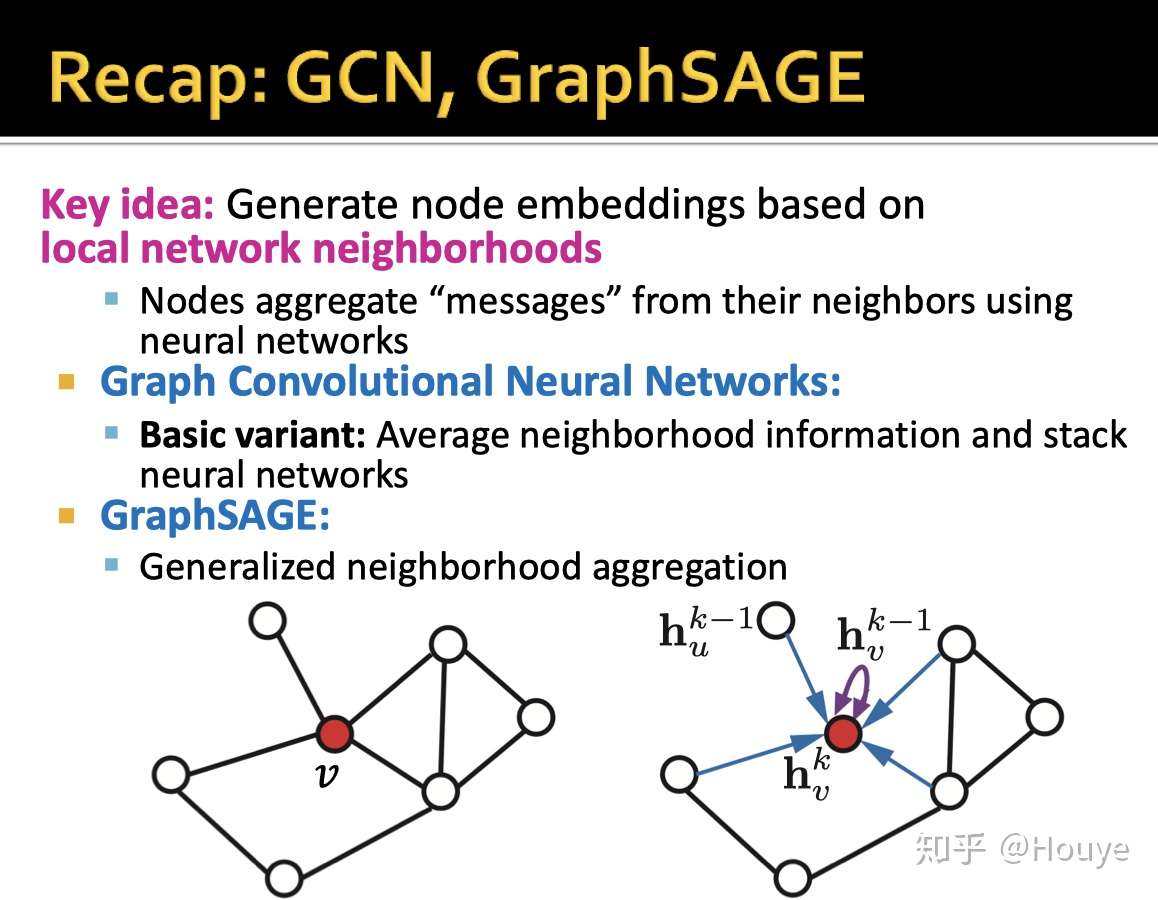
这里回顾了一下经典的GCN和GraphSAGE
"messages",即节点蕴含的信息.直接表示一个节点信息的就是其向量表示,也就是其node embedding.
CGN是中的消息传递(邻居聚合)对所有邻居进行平均.
GraphSAGE是中的消息传递(邻居聚合)对邻居进行采样,然后对一些邻居进行LSTM/Mean等聚合方式.
还有一种消息传递(邻居聚合)是对邻居加权聚合,可以识别邻居的重要性,过滤噪音邻居.比如,Graph Attention Network 和 Heterogeneous Graph Attention Network
回顾之前所谓的GNN,我们可以发现它们实际的encoder, follow都是这样的顺序:
图结构->gnn->节点表示.
本节的重点是图上的decoder. 与encoder相反, decoderfollow这样的顺序:
节点表示->decoder->图结构.
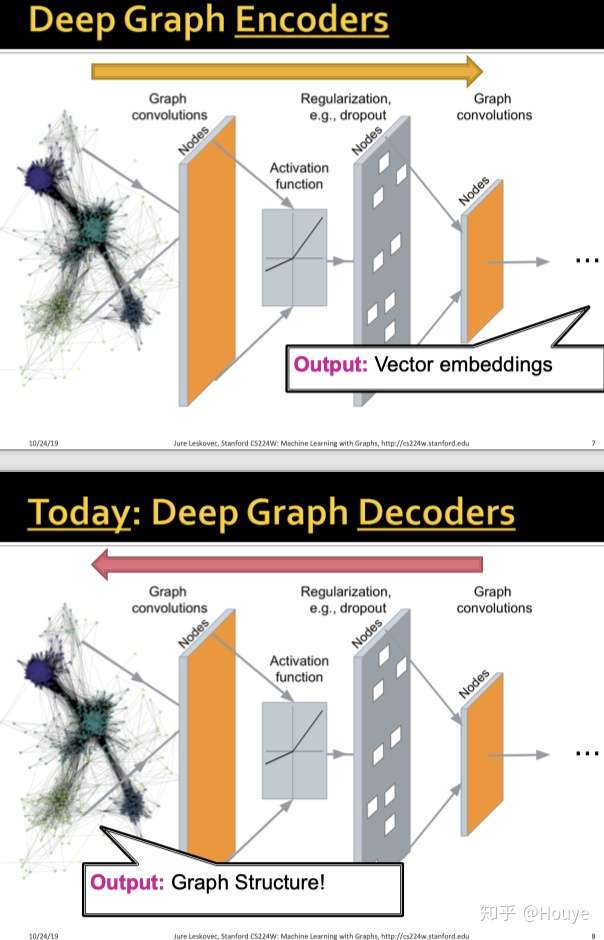
接下来是本节大纲

这里为什么要根据已有的图来生成新的图呢?
在药物化学领域,有一些很有效的分子结构具有一些我们希望的的性质,比如青霉素?
但是新药物分子的研发是非常费钱的. 看过<我不是药神>的同学应该都知道.
那么,能不能根据已有药物的分子结构,自动的去设计/发现新的药物分子结构呢?
这就对应到下图了. 旧的药物分子图->新的药物分子图.
接下来,Jure也会主要拿药物分子来做例子进行讲解.
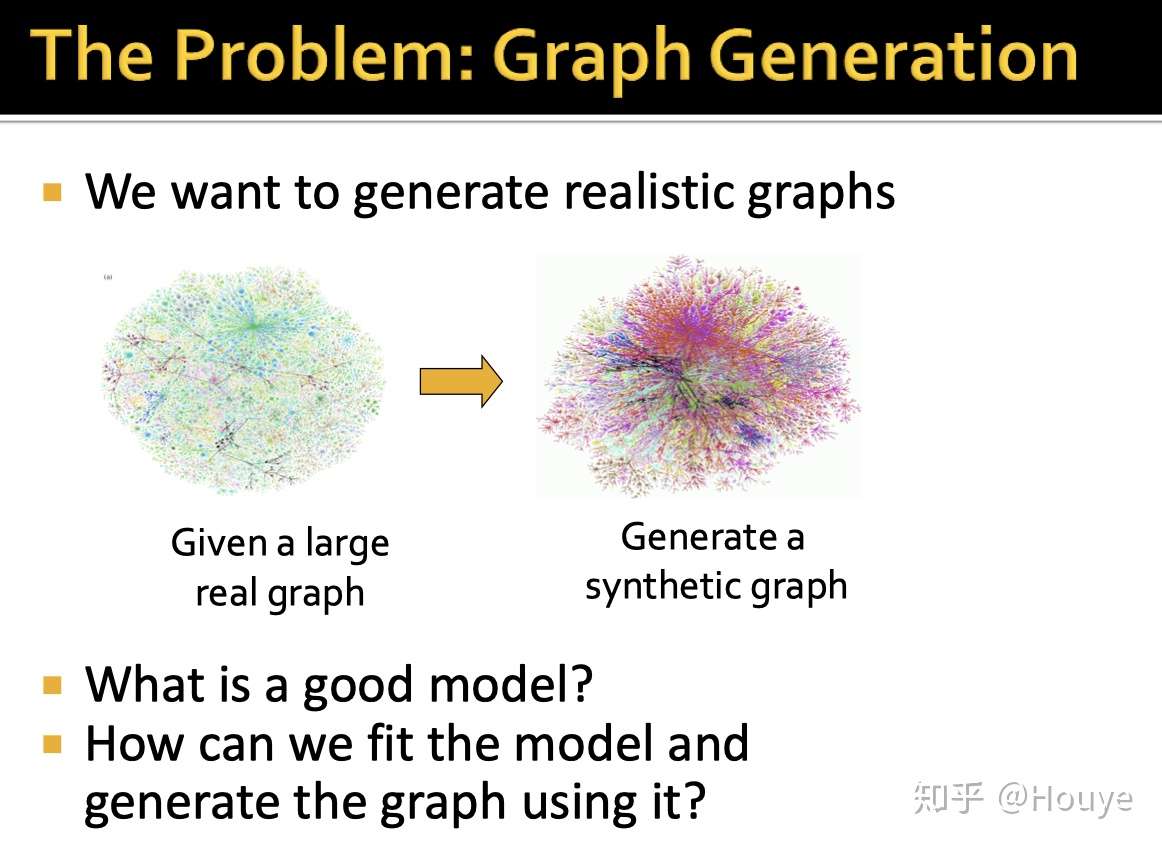
-下面是一些图生成的其他作用

-

-Graph Generative Model(GAM)除了可以直接生成新的药物分子外,
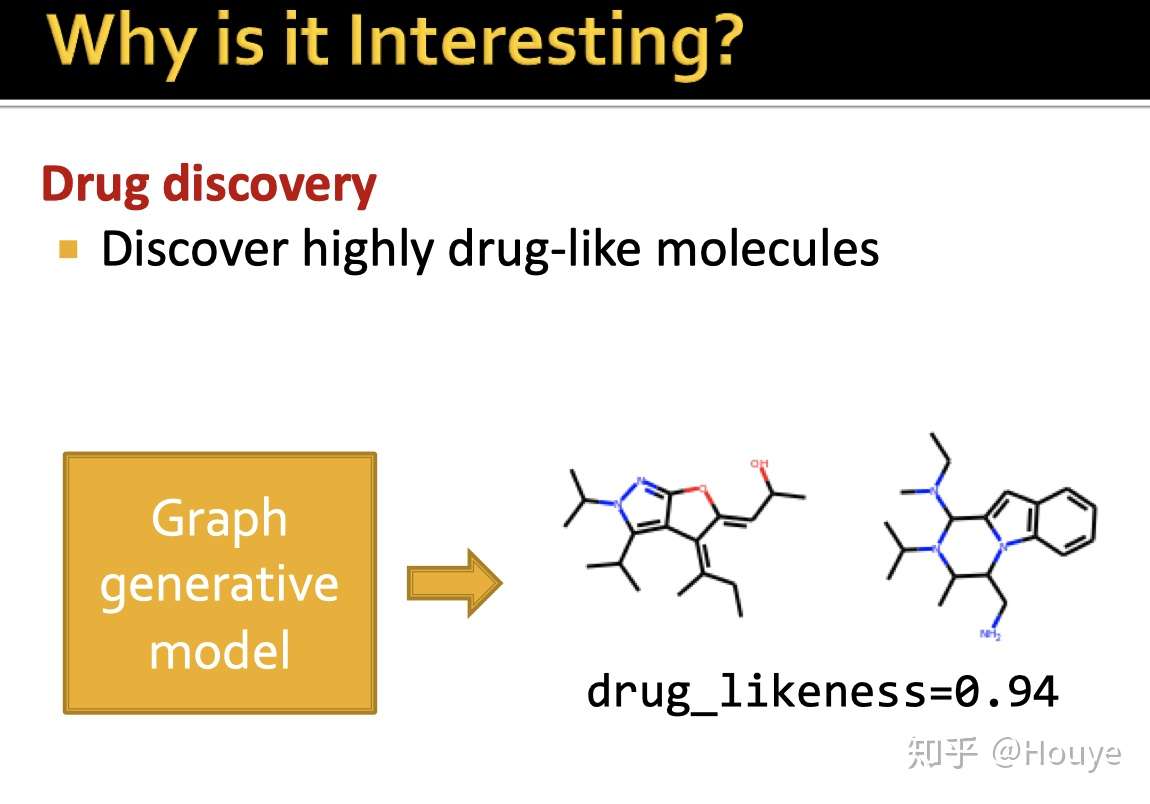
-GAM还可以对已有的药物分子进行拓展和补全,进一步提升其某些性质.
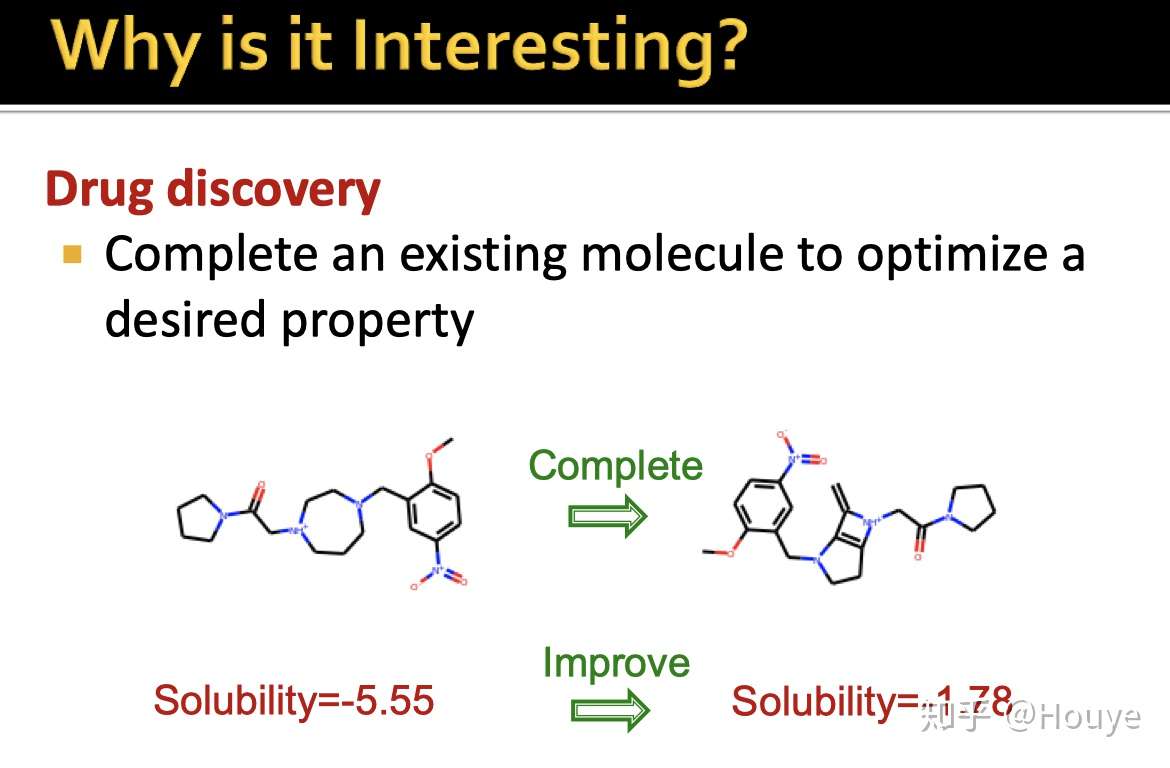
-除了具有极大经济价值的药物研发, GAM其实还有助于我们理解图本身的结构和性质.
这就类似于GAN,同为生成模型, 它们都可以更好的实现对data分布的描述.
毕竟你都可以生成数据了,必然对数据的所有特性了若指掌.
而不是像判别模型一样,只要抓住数据的某些特点就可以进行分类.
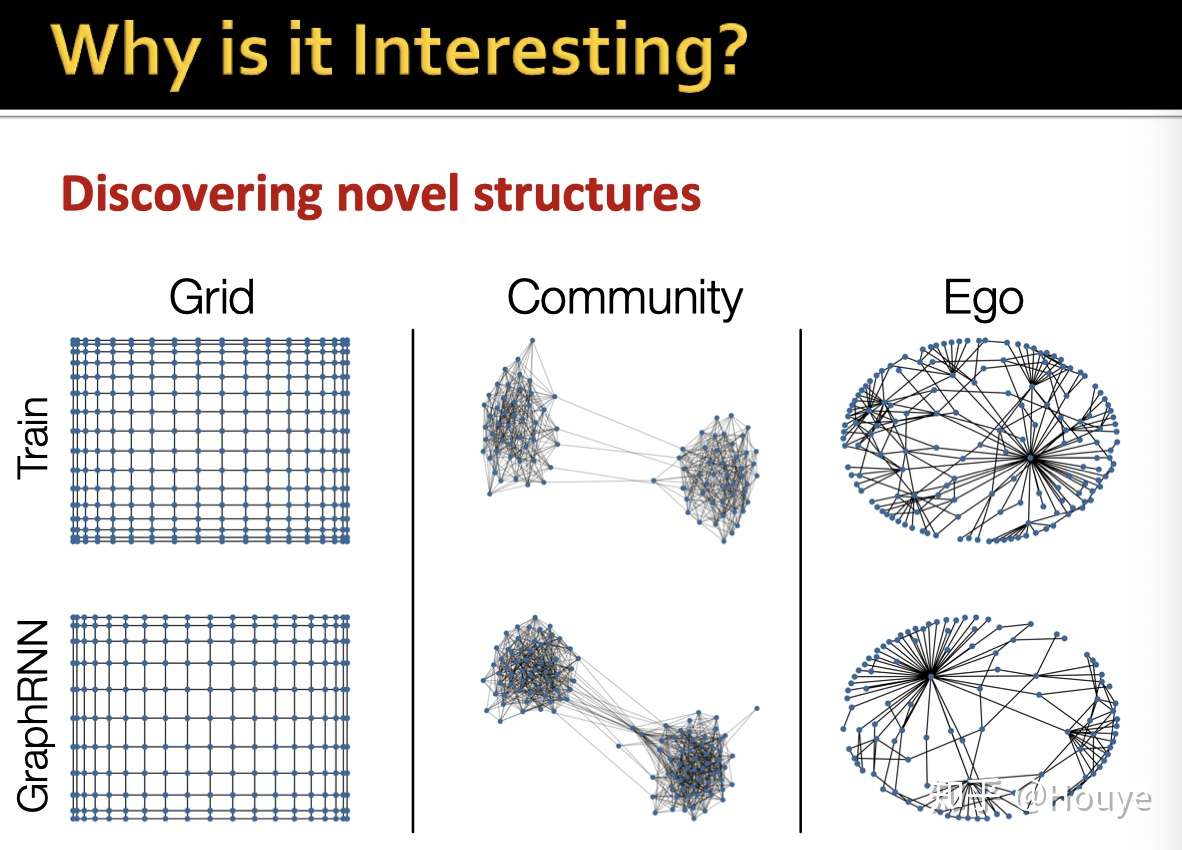
-既然GAM这么重要又非常有趣, 那么为什么大家还没做呢?
可能是因为GAM做起来太难了
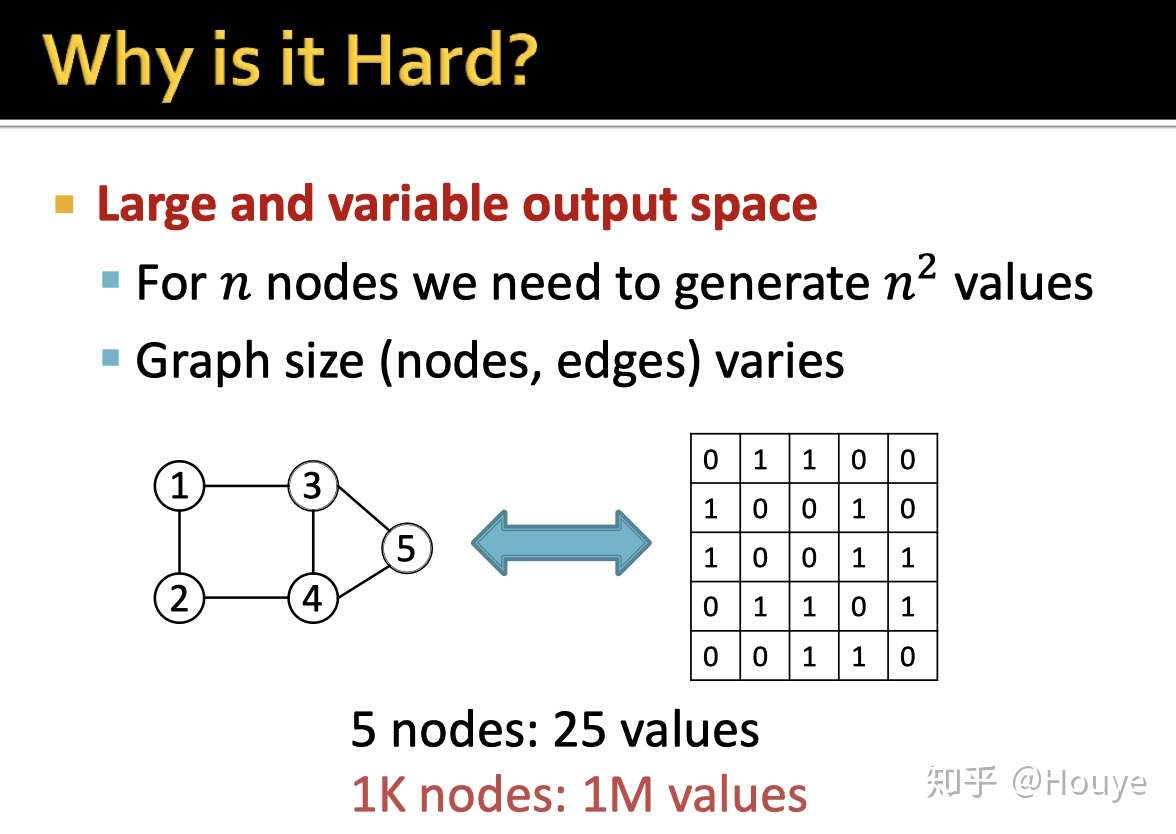
原因1: 输出的可能性太多(输出空间太大). 有n个节点的图,我们需要生成一个n*n的矩阵.
这个复杂度确实有点高.
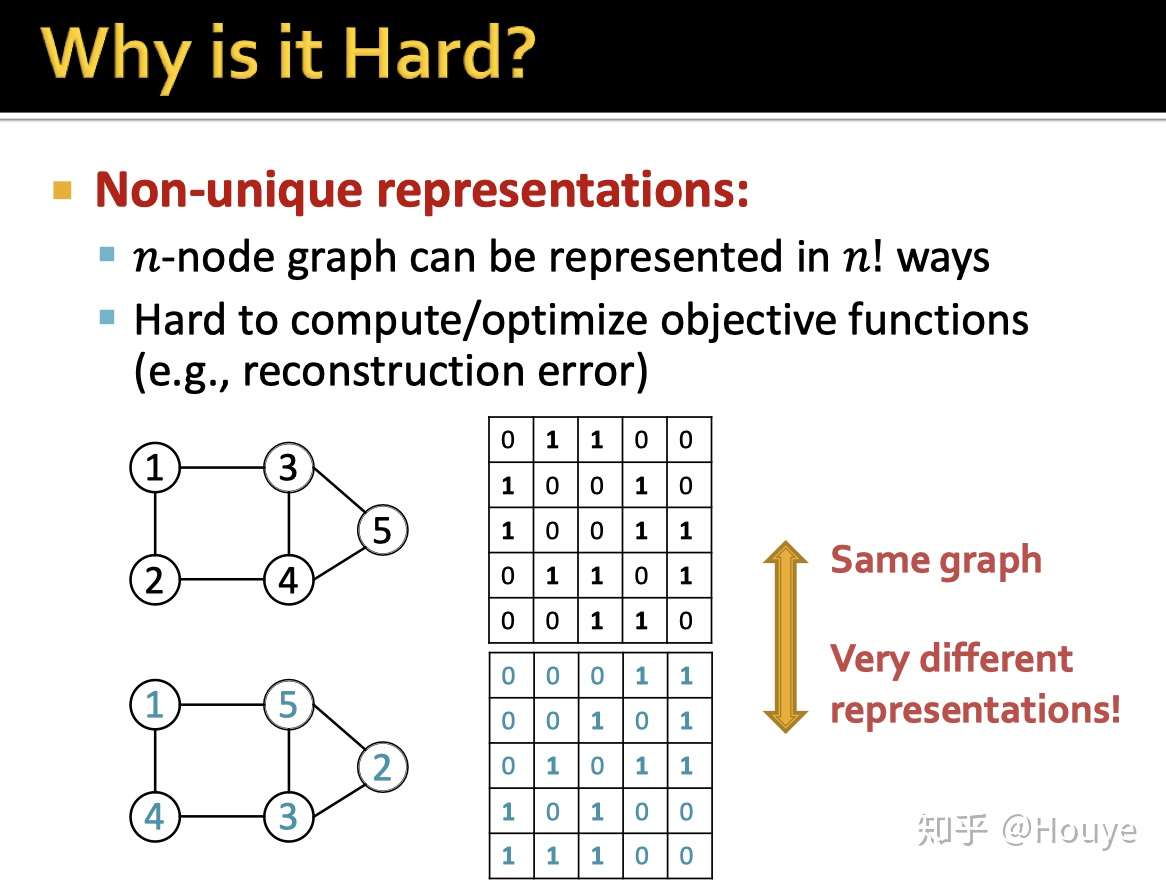
-原因2: 两张图,结构一模一样的,但是他们的邻居矩阵却可以不一样. 我们给n个节点编号,实际可能的编号方案有n!个.
-前面都在说复杂度的问题.
原因3: 图上两个节点之间是否存在边,其实除了考虑着两个节点之外,还应该更多的考虑高阶邻居的影响.
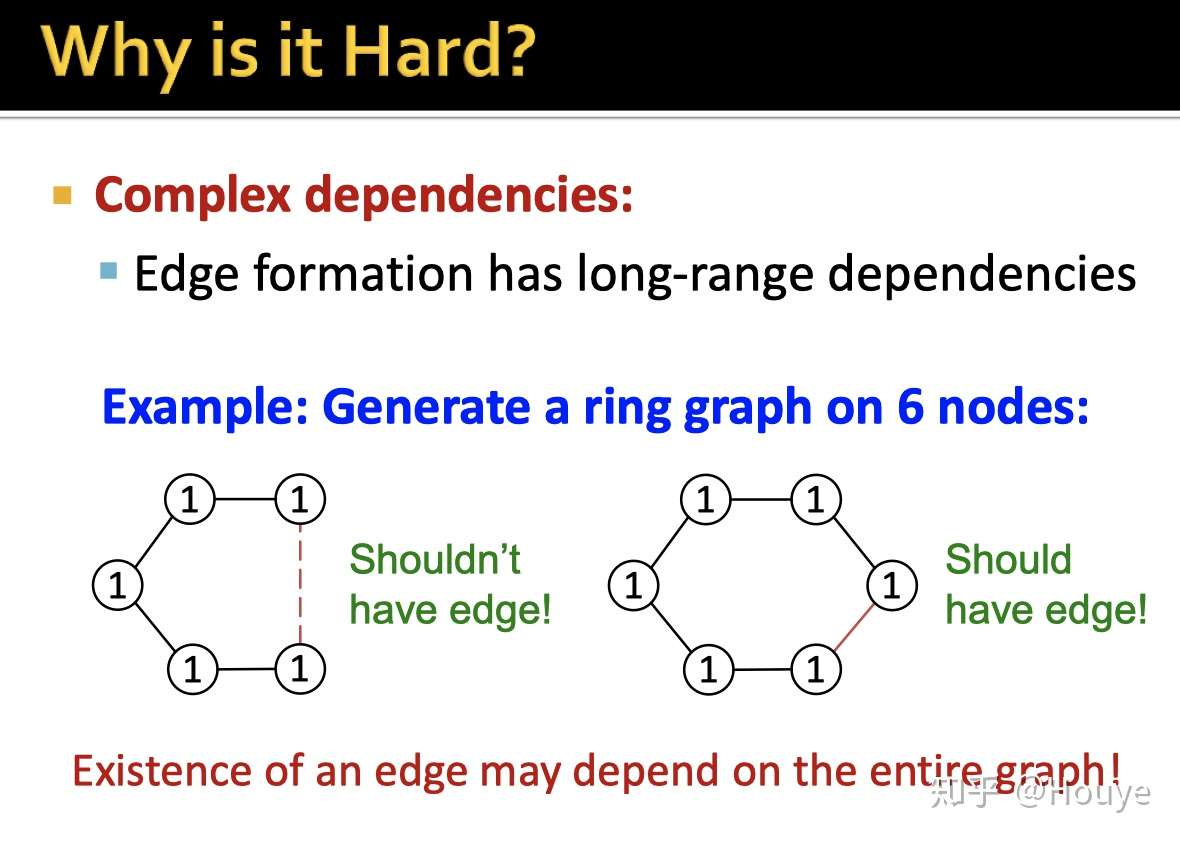
做过graph embedding的同学应该比较了解1阶邻居,2阶邻居,..., 高阶邻居.
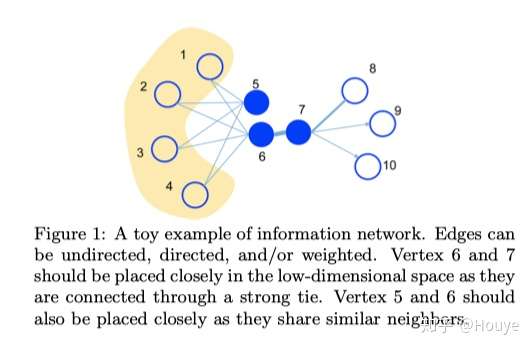
这里做一点点小小的补充. 下图来自15WWW LINE
如果需要预测5,6之间是否存在边,除了考虑它俩自身之外,其实1,2,3,4都应该被考虑进去.
前面都是一些通俗的介绍, 下面给一个图生成的形式化定义:

-先说p_data, 这里假定我们观测到的一系列图样本都是从某一个分布产生的.不同特点的图应该是不同的分布产生的.
我们的任务就是学一个分布p_model, 用model中大量参数去逼近真实的图分布p_data.也就是最小化distance(p_data, p_model)
然后,从分布采样就可以生成与现有图样本类似的新图了.
比如, 从一堆社交图学到的p_model会生成社交图.

回想这种参数估计的模型, 最经典的就是MLE. 只不过这里的每个样本是一个图.
当然这里也假设了数据是IID的

-这里是生成模型的典型做法
1.从一个正太分布中采样
2.对采样结果进行变换
比如下面链接里VAE
Houye:盘点深度学习中的不可导操作(次梯度和重参数化)zhuanlan.zhihu.com

这里, 分布学习和样本生成其实是两阶段的.
有没有一步到位为办法呢? Auro-regressive model就可以.
这里自回归的意思是:生成是按照顺序的;每一步都要综合考虑之前的所有步.
这里和前面所说的hard reason 3中节点间复杂长距离依赖其实是遥相呼应的.

编辑于 03-08 「一起学习图机器学习呀~」 还没有人赞赏,快来当第一个赞赏的人吧! 深度学习(Deep Learning) 图神经网络(GNN) 数据挖掘
文章被以下专栏收录
推荐阅读

CS224W 18-Limitations of Graph Neural Network
Houye发表于Houye...
ICLR20中15篇与图神经网络相关的论文:简单过一下
Mark发表于图网络学习...
GNN 简介和入门资料
钨额音
图神经网络的新基准Benchmarking Graph Neural Networks
Mark发表于图网络学习...1 条评论
写下你的评论...-
 John04-12
John04-12
请问这里讲的Graph Generative Models源自那篇paper?
标签:10.1,CS224W,药物,Models,Graph,生成,邻居,GNN,节点 来源: https://www.cnblogs.com/cx2016/p/13863757.html