R语言stan概率编程规划简介
作者:互联网
概率编程使我们能够实现统计模型,而无需担心技术细节。它对基于MCMC采样的贝叶斯模型特别有用。在本文中,我将研究如何通过在R 。
简介
RStan是贝叶斯推理的C ++库。它基于No-U-Turn采样器(NUTS),用于根据用户指定的模型和数据估计后验分布。使用Stan执行分析涉及以下步骤:
- 使用Stan建模语言指定统计模型。这通常通过专用的.stan文件完成。
- 准备要输入模型的数据。
- 使用该
stan函数从后验分布中取样。 - 分析结果。
在本文中,我将展示Stan使用两个分层模型的用法。我将使用第一个模型来讨论Stan的基本功能,并使用第二个示例来演示更高级的应用程序。
数据集
该数据集测量了教练计划对大学入学考试的影响,即美国使用的学术能力倾向测验(SAT)。SAT的设计应该能够抵抗直接针对提高测试成绩的短期努力。相反,测试应反映在较长时间内获得的知识。数据集如下:
| 学校 | 教练的估计效果(ÿĴÿĴ) | 标准效果误差(σĴσĴ) |
|---|---|---|
| a | 28 | 15 |
| b | 8 | 10 |
| C | -3 | 16 |
| d | 7 | 11 |
| e | -1 | 9 |
| f | 1 | 11 |
| g | 18 | 10 |
| h | 12 | 18 |
正如我们所看到的,国家税务总局没有履行承诺:对于八所学校中的大多数,短期教练确实增加了SAT成绩,正如 ÿĴÿĴ,哪里 ÿĴÿĴ表示SAT分数的变化。对于此数据集,我们有兴趣估计与每所学校相关的真实效果大小。可以使用两种替代方法。首先,我们可以假设所有学校都是相互独立的。然而,这将导致难以解释的估计,因为由于高标准误差,学校的95%后验间隔将在很大程度上重叠。其次,人们可以汇集所有学校的数据,假设所有学校的真实效果相同。然而,这也是不合理的,因为应该有针对学校的影响(例如不同的教师和学生)。
因此,需要另一种模型。分层模型的优点是结合了所有八所学校(实验)的信息,而没有假设它们具有共同的真实效果。我们可以通过以下方式指定层次贝叶斯模型
根据该模型,教练的效果遵循正态分布,其均值是真实效果, θĴθĴ,其标准差是 σĴσĴ,从数据中已知。真正的效果,θĴθĴ,遵循带参数的正态分布 μμ 和 ττ。
定义Stan模型文件
指定了我们将要使用的模型后,我们现在可以考虑如何在Stan中指定此模型。在为上面指定的模型定义Stan程序之前,让我们先看看Stan建模语言的结构。
变量
在Stan中,变量可以通过以下方式定义:
int<lower=0> n; # lower bound is 0
int<upper=5> n; # upper bound is 5
int<lower=0,upper=5> n; # n is in [0,5]
注意,如果它们是先验已知的,则应指定变量的上边界和下边界。
可以通过方括号指定多维数据:
vector[n] numbers; // a vector of length n
real[n] numbers; // an array of floats with length n
matrix[n,n] matrix; // an n times n matrix
程序块
Stan中使用了以下程序块:
- data:用于指定使用Bayes规则的条件
- 转换数据:用于预处理数据
- 参数(必需):用于指定模型的参数
- 变换后的参数:用于计算后验之前的参数处理
- model(必需):用于指定模型本身
- 生成数量:用于后处理结果
对于模型程序块,可以以两种等效方式指定分布。第一个,使用以下统计表示法:
y ~ normal(mu, sigma); # y follows a normal distribution
第二种方法使用基于对数概率密度函数(lpdf)的编程表示法:
target += normal_lpdf(y | mu, sigma); # increment the normal log density
Stan支持大量的概率分布。通过Stan指定模型时,该lookup功能派上用场:它提供了从R功能到Stan功能的映射。请考虑以下示例:
## StanFunction Arguments ReturnType Page
## 355 normal_rng (real mu, real sigma) real 494
在这里,我们看到rnormR 中的等价物是normal_rngStan。
八所学校的模型
现在我们已经了解了Stan建模langeu的基础知识,我们可以定义我们的模型,我们将它存储在一个名为的文件中schools.stan:
data {
int<lower=0> n; //number of schools
}
parameters {
vector[n] eta; // standardized school-level effects (see below)
}
transformed parameters {
}
model {
}
注意 θθ从未出现在参数中。这是因为我们没有明确建模θθ 而是模特 ηη,个别学校的标准化效果。然后我们构建θθ在变换后的参数部分中μμ, ττ,和 ηη。此参数化使采样器更有效。
请注意,模型是使用矢量符号指定的,因为两者都是 θθ 和 σσ表明向量。这样可以改善运行时间,因为不需要遍历每个单独的元素。
准备数据进行建模
在我们拟合模型之前,我们需要将输入数据编码为一个列表,其参数应该与Stan模型的数据部分中的条目相对应。对于学校数据,数据如下:
从后验分布中取样
我们可以使用stan函数从后验分布中进行采样,执行以下三个步骤:
- 它将模型规范转换为C ++代码。
- 它将C ++代码编译为共享对象。
- 它根据指定的模型,数据和设置从后验分布中进行采样。
如果rstan_options(auto_write = TRUE)已在活动R会话中执行,则相同模型的后续调用将比第一次调用快得多,因为该stan函数然后跳过前两个步骤(转换和编译模型)。此外,我们将设置要使用的核心数:
现在,我们可以从后验编译模型和样本。唯一需要的两个参数stan是模型文件的位置和要输入模型的数据。此处显示的其他参数仅用于提供可能选项的概述:
模型解释
我们将首先对模型进行基本解释,然后研究MCMC程序。
基本模型解释
要使用拟合模型进行推理,我们可以使用该print函数。
## Inference for Stan model: schools.
## 4 chains, each with iter=2000; warmup=1000; thin=1;
## post-warmup draws per chain=1000, total post-warmup draws=4000.
##
## mean se_mean sd 2.5% 25% 50% 75% 97.5% n_eff Rhat
## mu 7.67 0.15 5.14 -2.69 4.42 7.83 10.93 17.87 1185 1
## tau 6.54 0.16 5.40 0.31 2.52 5.28 9.05 20.30 1157 1
## eta[1] 0.42 0.01 0.92 -1.47 -0.18 0.44 1.03 2.18 4000 1
## eta[2] 0.03 0.01 0.87 -1.74 -0.54 0.03 0.58 1.72 4000 1
## eta[3] -0.18 0.02 0.92 -1.95 -0.81 -0.20 0.45 1.65 3690 1
## eta[4] -0.03 0.01 0.92 -1.85 -0.64 -0.02 0.57 1.81 4000 1
## eta[5] -0.33 0.01 0.86 -2.05 -0.89 -0.34 0.22 1.43 3318 1
## eta[6] -0.20 0.01 0.87 -1.91 -0.80 -0.21 0.36 1.51 4000 1
## eta[7] 0.37 0.02 0.87 -1.37 -0.23 0.37 0.96 2.02 3017 1
## eta[8] 0.05 0.01 0.92 -1.77 -0.55 0.05 0.69 1.88 4000 1
## theta[1] 11.39 0.15 8.09 -2.21 6.14 10.30 15.56 30.22 2759 1
## theta[2] 7.92 0.10 6.25 -4.75 4.04 8.03 11.83 20.05 4000 1
## theta[3] 6.22 0.14 7.83 -11.41 2.03 6.64 10.80 20.97 3043 1
## theta[4] 7.58 0.10 6.54 -5.93 3.54 7.60 11.66 20.90 4000 1
## theta[5] 5.14 0.10 6.30 -8.68 1.40 5.63 9.50 16.12 4000 1
## theta[6] 6.08 0.10 6.62 -8.06 2.21 6.45 10.35 18.53 4000 1
## theta[7] 10.60 0.11 6.70 -0.94 6.15 10.01 14.48 25.75 4000 1
## theta[8] 8.19 0.14 8.18 -8.13 3.59 8.01 12.48 25.84 3361 1
## lp__ -39.47 0.07 2.58 -45.21 -41.01 -39.28 -37.70 -34.99 1251 1
##
## Samples were drawn using NUTS(diag_e) at Thu Nov 29 11:17:50 2018.
## For each parameter, n_eff is a crude measure of effective sample size,
## and Rhat is the potential scale reduction factor on split chains (at
## convergence, Rhat=1).
这里,行名称表示估计的参数:mu是后验分布的均值,tau是其标准偏差。eta和theta的条目表示向量的估计ηη 和 θθ, 分别。该LP条目显示日志密度,这通常是不相关的。列指示计算的值。百分比表示可信区间。例如,教练整体效果的95%可信区间,μμ是的 [ - 1.27 ,18.26 ][- 1.27,18.26]。由于我们不太确定平均值,95%的可信区间为θĴθĴ也很宽。例如,对于第一所学校,95%的可信区间是[ - 2.19 ,32.33 ][- 2.19,32.33]。
我们可以使用plot函数可视化估算中的不确定性:
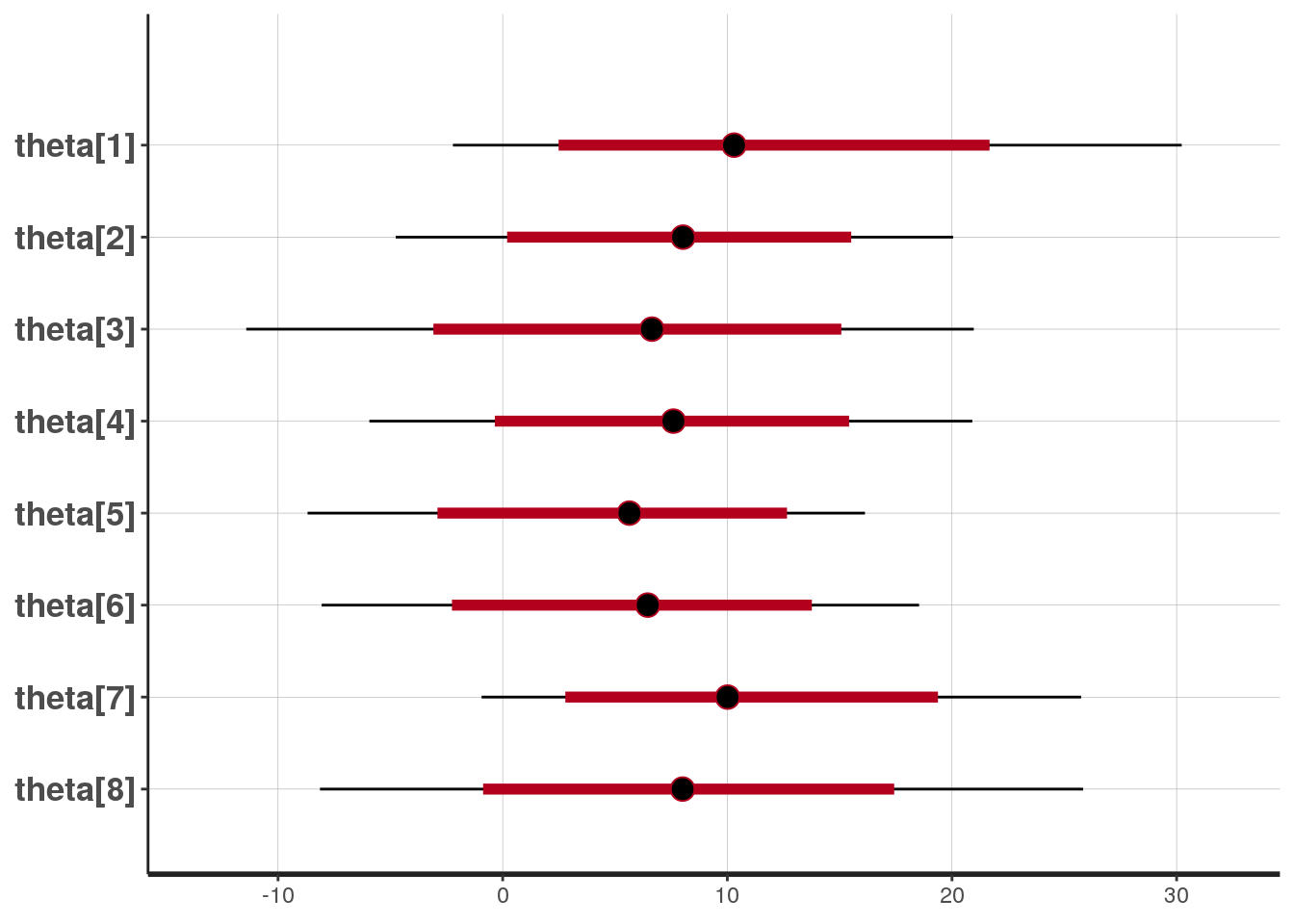


黑线表示95%的间隔,而红线表示80%的间隔。圆圈表示平均值的估计值。
我们可以使用以下extract函数获取生成的样本:
MCMC诊断
通过绘制采样程序的轨迹,我们可以确定采样过程中是否出现任何问题。例如,如果链条在一个地方停留太长时间或在一个方向上形成太多步骤,则可能是这种情况。我们可以使用traceplot函数绘制模型中使用的四条链的痕迹:
<span style="color:#000000"><span style="color:#000000"><code><span style="color:#888888"># diagnostics:</span>
traceplot(fit1, pars = c(<span style="color:#880000">"mu"</span>, <span style="color:#880000">"tau"</span>), inc_warmup = <span style="color:#78a960">TRUE</span>, nrow = <span style="color:#880000">2</span>)</code></span></span>

两条痕迹对我来说都很好。
为了从单个马尔可夫链获得样本,我们可以extract再次使用该函数:
## parameters
## chains mu tau eta[1] eta[2] eta[3] eta[4]
## chain:1 1.111120 2.729124 -0.1581242 -0.8498898 0.5025965 -1.9874554
## chain:2 3.633421 2.588945 1.2058772 -1.1173221 1.4830778 0.4838649
## chain:3 13.793056 3.144159 0.6023924 -1.1188243 -1.2393491 -0.6118482
## chain:4 3.673380 13.889267 -0.0869434 1.1900236 -0.0378830 -0.2687284
## parameters
## chains eta[5] eta[6] eta[7] eta[8] theta[1]
## chain:1 0.3367602 -1.1940843 0.5834020 -0.08371249 0.6795797
## chain:2 -1.8057252 0.7429594 0.9517675 0.55907356 6.7553706
## chain:3 -1.5867789 0.6334288 -0.4613463 -1.44533007 15.6870727
## chain:4 0.1028605 0.3481214 0.9264762 0.45331024 2.4657999
## parameters
## chains theta[2] theta[3] theta[4] theta[5] theta[6] theta[7]
## chain:1 -1.208335 2.482769 -4.31289292 2.030181 -2.147684 2.703297
## chain:2 0.740736 7.473028 4.88612054 -1.041502 5.556902 6.097494
## chain:3 10.275294 9.896345 11.86930758 8.803971 15.784656 12.342510
## chain:4 20.201935 3.147213 -0.05906019 5.102037 8.508530 16.541455
## parameters
## chains theta[8] lp__
## chain:1 0.8826584 -41.21499
## chain:2 5.0808317 -41.17178
## chain:3 9.2487083 -40.35351
## chain:4 9.9695268 -36.34043
为了对采样过程进行更高级的分析,我们可以使用shinystan提供Shiny前端的包。使用该软件包,可以通过以下方式启动Shiny应用程序来分析拟合模型:
分层回归
现在我们对Stan有了基本的了解,我们可以深入了解更高级的应用程序:让我们尝试分层回归。在传统的回归中,我们模拟了形式的关系
ÿ= β0+ X.β。ÿ=β0+Xβ。
该表示假定所有样本具有相同的分布。如果存在一组样本,那么我们就会遇到问题,因为组内和组之间的潜在差异将被忽略。
另一种方法是为每个组建立一个回归模型。然而,在这种情况下,在估计单个模型时,小样本量将是有问题的。
分层回归是两个极端之间的折衷。该模型假设这些组相似但仍然表现出差异。
假设每个样本属于其中一个 ķķ组。然后,分层回归指定如下:
ÿķ= αķ+ X.ķβ(k ),∀ ķ ∈ { 1 ,... ,ķ}ÿķ=αķ+Xķβ(ķ),∀ķ∈{1,...,ķ}
哪里 ÿķÿķ 是结果 ķķ- 小组, αķαķ 是拦截, XķXķ 是功能,和 β(k )β(ķ)表示权重。层次模型与模型不同ÿķÿķ 因为参数适合每个组, αķαķ 和 β(k )β(ķ) 假定来自共同分布。
大鼠数据集
分层回归的典型示例是大鼠数据集。该纵向数据集包含测量5周的大鼠重量。让我们加载数据:
## day8 day15 day22 day29 day36
## 1 151 199 246 283 320
## 2 145 199 249 293 354
## 3 147 214 263 312 328
## 4 155 200 237 272 297
## 5 135 188 230 280 323
## 6 159 210 252 298 331

数据显示出不同大鼠的线性增长趋势非常相似。然而,我们也看到大鼠具有不同的初始重量,这需要不同的截距,以及不同的生长速率,这需要不同的斜率。因此,分层模型似乎是合适的。
分层回归模型的规范
我们现在可以指定模型并将其存储在一个名为的文件中rats.stan:
data {
int<lower=0> N; // the number of rats
int<lower=0> T; // the number of time points
real y[N,T]; // matrix of weight times time
real xbar; // the median number of days in the time series
}
parameters {
real alpha[N]; // the intercepts of rat weights
real mu_alpha; // the mean intercept
real mu_beta; // the mean slope
real<lower=0> sigmasq_y;
real<lower=0> sigmasq_alpha;
}
transformed parameters {
real<lower=0> sigma_y; // sd of rat weight
real<lower=0> sigma_alpha; // sd of intercept distribution
sigma_y <- sqrt(sigmasq_y);
sigma_alpha <- sqrt(sigmasq_alpha);
sigma_beta <- sqrt(sigmasq_beta);
}
model {
mu_alpha ~ normal(0, 100); // non-informative prior
mu_beta ~ normal(0, 100); // non-informative prior
sigmasq_alpha ~ inv_gamma(0.001, 0.001); // conjugate prior of normal
sigmasq_beta ~ inv_gamma(0.001, 0.001); // conjugate prior of normal
for (n in 1:N) // for each sample
for (t in 1:T) // for each time point
y[n,t] ~ normal(alpha[n] + beta[n] * (x[t] - xbar), sigma_y);
}
generated quantities {
// determine the intercept at time 0 (birth weight)
alpha0 <- mu_alpha - xbar * mu_beta;
}
请注意,模型代码估计方差(sigmasq变量)而不是标准偏差。此外,生成的数量块显式计算α0α0,时间0的截距,即出生时大鼠的体重。我们还可以计算生成量块中的任何其他数量,例如,不同时间点的大鼠的估计重量。我们稍后会在R中执行此操作。
数据准备
要为模型准备数据,我们首先将测量点提取为数值,然后在列表结构中对所有内容进行编码:
拟合回归模型
我们现在可以拟合大鼠体重数据集的贝叶斯分层回归模型:
用层次回归模型预测
确定了 αα 和 ββ对于每只大鼠,我们现在可以在任意时间点估计个体大鼠的体重。在这里,我们感兴趣的是从第0天到第100天找到大鼠的体重。



与原始数据相比,模型的估计是平滑的,因为每条曲线都遵循线性模型。研究最后一个图中显示的置信区间,我们可以看出方差估计是合理的。我们对采样时的大鼠重量(第8天到第36天)充满信心,但随着我们离采样区域越远,不确定性越大。
如果您有任何疑问,请在下面发表评论。
标签:chain,##,简介,模型,编程,Stan,eta,stan,theta 来源: https://blog.csdn.net/qq_19600291/article/details/89888740